NEWS
Corona-Daten nach MySQL importieren
-
@sissiwup cool, das sieht schon besser aus.
Jetzt meckert grafana über eine Tabelle datapoints, die sie nicht finden kann. Kannst du auch hier helfen?
Erledigt. Das lag daran, dass ich in der DB nur deine Daten habe und die aus iobroker nicht. -
Die csv-Datei für rki ist nicht mehr komplett ladbar (vermutlich ein Fehler auf der Seite)
PS: Datenstand in der cor_rki Tabelle muss 22 Zeichen lang sein...
Hier eine kleine Umgehungslösung:
rkijson.pyimport csv, json import datetime as dt import requests outfile = r'/var/skripte/data/cor_rki.csv' count = "https://services7.arcgis.com/mOBPykOjAyBO2ZKk/arcgis/rest/services/RKI_COVID19/FeatureServer/0/query?where=1%3D1&outFields=*&returnCountOnly=true&f=pjson" anz = requests.get(count) anz_json = json.loads(anz.text) anzahl = anz_json['count'] print("Zeilen:" + str(anzahl)) ofile = open(outfile, 'w+') output = csv.writer(ofile) search1 = "https://services7.arcgis.com/mOBPykOjAyBO2ZKk/arcgis/rest/services/RKI_COVID19/FeatureServer/0/query?where=1%3D1&outFields=*&resultOffset=" search2 = "&resultRecordCount=1000&f=pjson" i = 0 while i < anzahl: print("Readfile:" + str((int)(i / 1000)) + " von " + str((int)(int(anzahl) / 1000))) s_str = search1 + str(i) + search2 x = requests.get(s_str) data = json.loads(x.text) daten = data['features'] for row in daten: zeit = row['attributes']['Meldedatum'] / 1000 row['attributes']['Meldedatum'] = dt.datetime.utcfromtimestamp(zeit).strftime( "%Y-%m-%dT%H:%M:%S.000Z") if i == 0: output.writerow(daten[0]['attributes'].keys()) for row in daten: output.writerow(row['attributes'].values()) i = i + 1000Das Downloadskript sieht dann wie folgt aus:
#!/bin/bash NOW=`date +"%d.%m.%g %H:%M.%S"` NOWDAT=`date +"%d_%m_%g"` USER=DBUSER PASS=DBPASSWORT rm /var/skripte/data/cor*.csv #wget -O /var/skripte/data/cor_rki.csv https://opendata.arcgis.com/datasets/dd4580c810204019a7b8eb3e0b329dd6_0.csv python3 -u /var/skripte/rkijson.py wget -O /var/skripte/data/cor_landkreise.csv https://opendata.arcgis.com/datasets/917fc37a709542548cc3be077a786c17_0.csv wget -O /var/skripte/data/cor_bundesland.csv https://opendata.arcgis.com/datasets/ef4b445a53c1406892257fe63129a8ea_0.csv cp /var/skripte/data/cor_rki.csv /var/skripte/data/rki_$NOWDAT.csv.backup cp /var/skripte/data/cor_landkreise.csv /var/skripte/data/landkreise_$NOWDAT.csv.backup cp /var/skripte/data/cor_bundesland.csv /var/skripte/data/bundesland_$NOWDAT.csv.backup #mysql -u $USER -p$PASS iobroker < /var/skripte/data/createTable.txt mysqlimport --fields-terminated-by=, --ignore-lines=1 --verbose --delete --local -u $USER -p$PASS iobroker /var/skripte/data/cor_rki.csv mysqlimport --fields-terminated-by=, --ignore-lines=1 --verbose --delete --local -u $USER -p$PASS iobroker /var/skripte/data/cor_landkreise.csv mysqlimport --fields-terminated-by=, --ignore-lines=1 --verbose --delete --local -u $USER -p$PASS iobroker /var/skripte/data/cor_bundesland.csv mysql -u $USER -p$PASS iobroker < /var/skripte/data/createZiel.txt -
Hier noch die JHU Daten:
Skript:
#!/bin/bash NOW=`date +"%d.%m.%g %H:%M.%S"` NOWDAT=`date +"%d_%m_%g"` USER=DBUSER PASS=DBPASSWORT rm /var/skripte/data/jhu*.csv wget -O /var/skripte/data/jhu_fall.csv https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/time_series_covid19_confirmed_ global.csv wget -O /var/skripte/data/jhu_tote.csv https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/time_series_covid19_deaths_glo bal.csv wget -O /var/skripte/data/jhu_genesen.csv https://raw.githubusercontent.com/CSSEGISandData/COVID-19/master/csse_covid_19_data/csse_covid_19_time_series/time_series_covid19_recover ed_global.csv cp /var/skripte/data/jhu_fall.csv /var/skripte/data/fall_$NOWDAT.csv.backup cp /var/skripte/data/jhu_tote.csv /var/skripte/data/tote_$NOWDAT.csv.backup cp /var/skripte/data/jhu_genesen.csv /var/skripte/data/genesen_$NOWDAT.csv.backup python3 -u /var/skripte/convertJHU.py mysql -u $USER -p$PASS iobroker < /var/skripte/data/createJHU.txt mysqlimport --fields-terminated-by=, --ignore-lines=1 --verbose --delete --local -u $USER -p$PASS iobroker /var/skripte/data/cor_jhu.csv mysql -u $USER -p$PASS iobroker < /var/skripte/data/updateJHU.txtund das convertJHU.py
import csv import datetime as dt outfile = r'/var/skripte/data/cor_jhu.csv' infile1 = r'/var/skripte/data/jhu_fall.csv' infile2 = r'/var/skripte/data/jhu_genesen.csv' infile3 = r'/var/skripte/data/jhu_tote.csv' now = dt.date.today() def conv_date(ind): val = ind.split("/") txt = "20" + val[2] + "-" + ("0" + val[0])[-2:] + "-" + ("0" + val[1])[-2:] return txt def read_files(csv_reader, fall_art): for row in csv_reader: keys = list(row.keys()) for subkey in keys[4:]: if row["Country/Region"] == "Canada": row["Province/State"] = "" nice_key = conv_date(subkey) key = nice_key + ":" + row["Country/Region"] + ":" + row["Province/State"] # print("Key=" + key) if key not in countrys.keys(): lists = {"Meldedatum": nice_key, "Land": row["Country/Region"], "Bundesland": row["Province/State"], "Import": now, fall_art: row[subkey]} # print("List=" + str(lists)) countrys[key] = lists # print("C=" + str(countrys)) else: lists = countrys[key] if fall_art not in lists.keys(): lists[fall_art] = row[subkey] else: lists[fall_art] = row[subkey] + lists[fall_art] countrys[key] = lists countrys = {} in_f = open(infile1) csv_reader = csv.DictReader(in_f) read_files(csv_reader, "fall") in_f = open(infile2) csv_reader = csv.DictReader(in_f) read_files(csv_reader, "genesen") in_f = open(infile3) csv_reader = csv.DictReader(in_f) read_files(csv_reader, "tote") print(len(countrys)) # print(countrys) # for row in list(countrys.values())[:10]: # print(row) out_f = open(outfile, "w+") fields = ["Meldedatum", "Land", "Bundesland", "fall", "genesen", "tote", "fall_tag" ,"genesen_tag" ,"tote_tag" ,"Import"] csv_writer = csv.DictWriter(out_f, fieldnames=fields) csv_writer.writerow(dict((fn, fn) for fn in fields)) for row in list(countrys.values())[:]: csv_writer.writerow(row)Canada wird gesondert
behandelt, da genesene nur für Gesamtcanada vorhanden sind.Und die Tabelle createJHU.txt:
CREATE TABLE IF NOT EXISTS cor_jhu( Meldedatum varchar(10) NOT NULL, Land varchar(50) NOT NULL, Bundesland varchar(50) DEFAULT NULL, fall int(11) NOT NULL, genesen int(11) NOT NULL, tote int(11) NOT NULL, fall_tag int(11) NOT NULL, genesen_tag int(11) NOT NULL, tote_tag int(11) NOT NULL, Import varchar(10) NOT NULL ) DEFAULT CHARACTER SET = UTF8; ALTER TABLE `cor_jhu` ADD UNIQUE KEY `U1` (`Meldedatum`,`Land`,`Bundesland`), ADD UNIQUE KEY `U2` (`Land`,`Bundesland`,`Meldedatum`), ADD KEY `I1` (`Land`,`Meldedatum`,`fall`), ADD KEY `I2` (`Land`,`Meldedatum`,`genesen`), ADD KEY `I3` (`Land`,`Meldedatum`,`tote`); COMMIT; truncate cor_jhu;Und die SQL-Korrektur updateJHU.txt:
update cor_jhu set land="Korea",bundesland="South" where land like "%Korea%" and Bundesland like "%South%"; update cor_jhu j1,cor_jhu j2 set j1.fall_tag=j1.fall-j2.fall,j1.tote_tag=j1.tote-j2.tote,j1.genesen_tag=j1.genesen-j2.genesen WHERE j1.land=j2.land and j1.bundesland=j2.bundesland and date_sub(j1.meldedatum,interval 1 day)=j2.meldedatum and j1.meldedatum>date("2020-01-22"); COMMIT -
Hier mal ein Vergleich JHU und RKI:
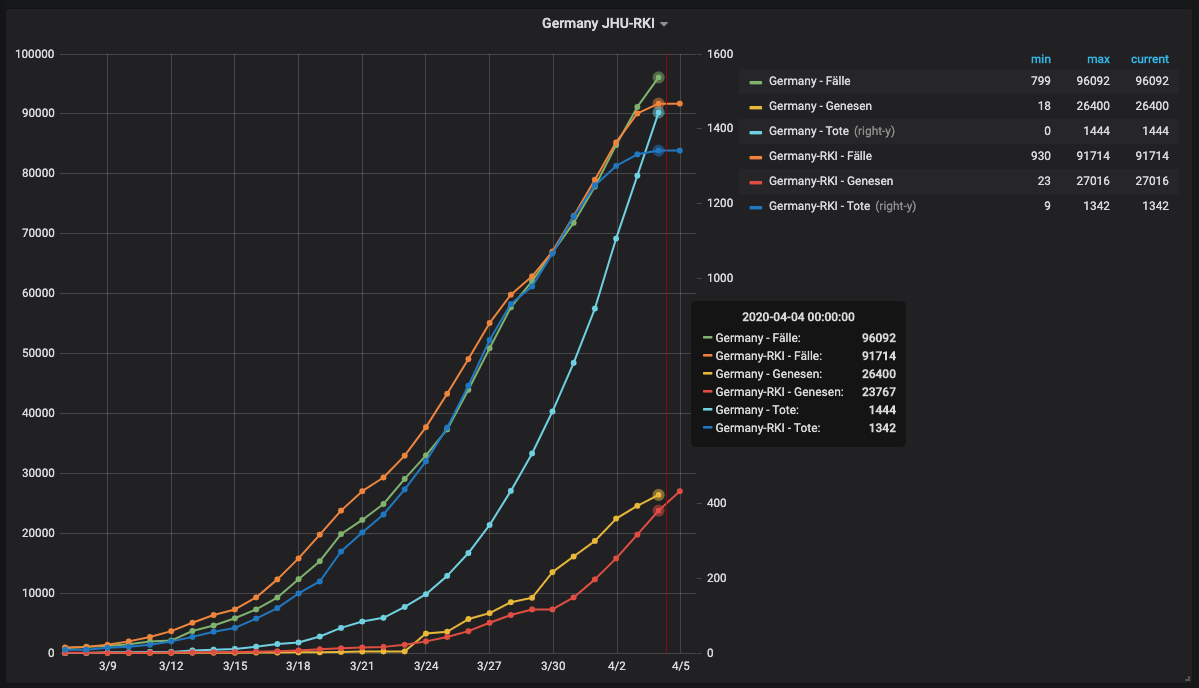
-
Hallo,
und hier mal ein paar Auswertungen auf den JHU_daten:
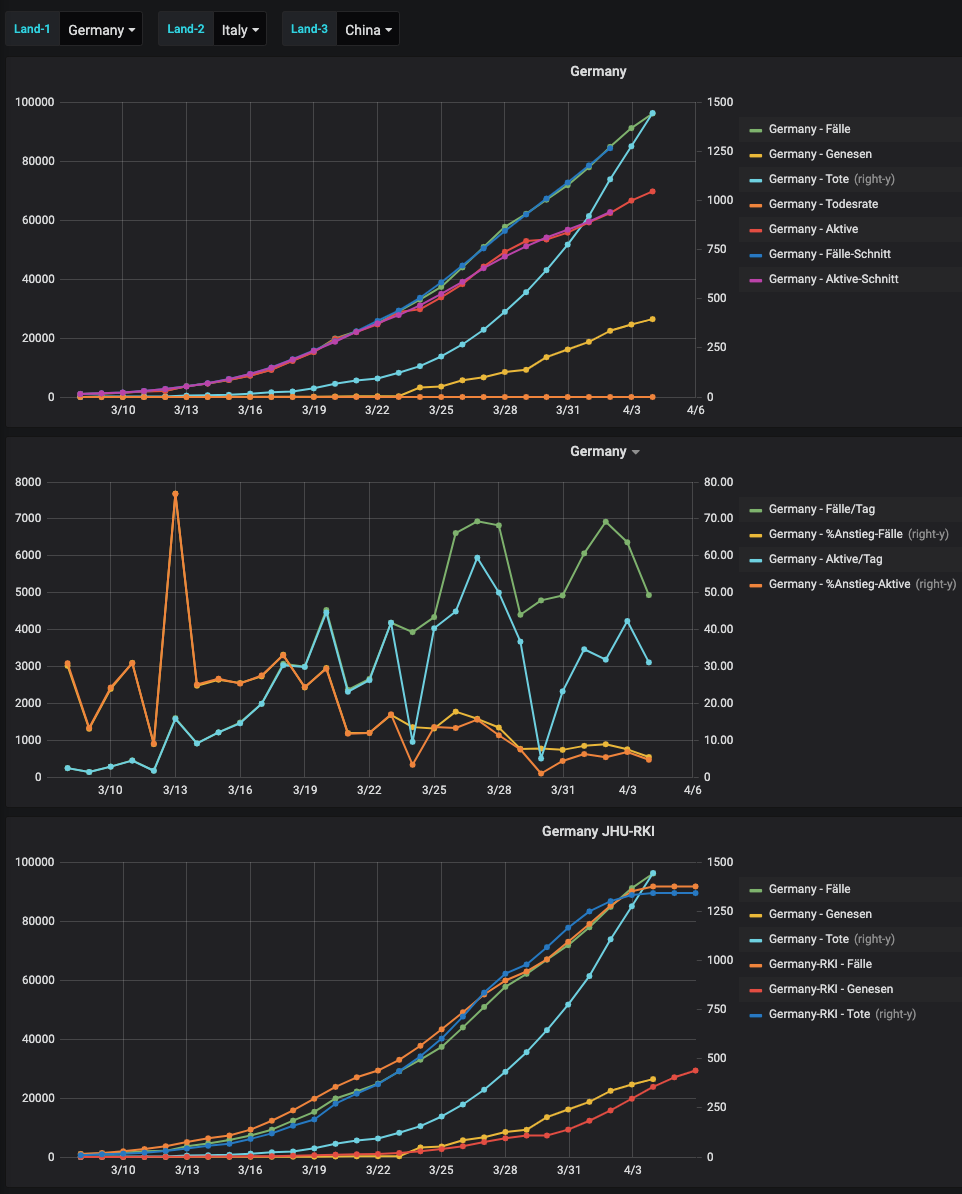
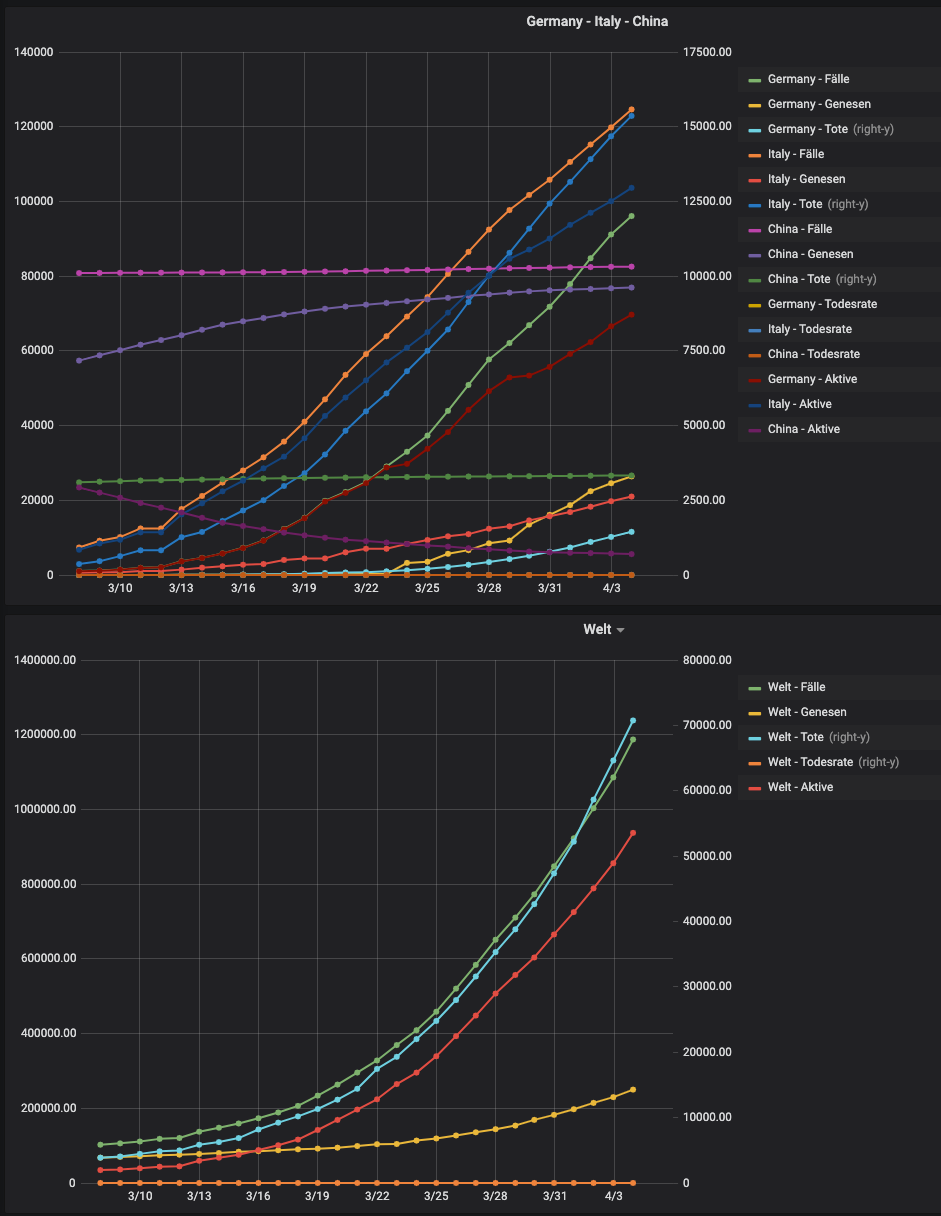
-
@sissiwup und Kollegen!
Bin jetzt 2 in Quarantäne gewesen und seit dem Wochenende zu Hause.
Hatte nur ein altes Chrome-book mit mir welches keine linux-Befehle erlaubte und einen neuen 4GB Raspi den ich mir vorher noch gekauft hatte. Ich habe wegen meiner fehlenden Testmöglichkeiten kein iobroker-adapter geschrieben aber eine kleine web-app mit der die Krankheitsverlaufkurve von Staaten angezeigt und verglöichen werden kann.
Ihr könnt die App direkt auf git anschauen: https://frankjoke.github.io/coronafj/
Das repo dazu ist https://github.com/frankjoke/coronafjEs gibt einen chart tab und einen list tab, sonst sollte es selbsterklärend sein.
Da ich das für meine Kollegen aus allen Ländern gemacht habe ist's halt in Englisch, aber man kann sehr schön die Unterschiedliche Entwicklung der Länder sehen!Übrigens, bin ab jetzt in Pension (oder Rente wie ihr sagen würdet) und hoffe mich bald wieder meinen iobroker-Adaptern widmen zu können, wir dürfen sowieso nicht wirklich rausgehen hier in A!
p.s.: verwende eine api verwendet die von da https://gisanddata.maps.arcgis.com/apps/opsdashboard/index.html#/bda7594740fd40299423467b48e9ecf6 gespeist wird.
-
@frankjoke sagte in Corona-Daten nach MySQL importieren:
Übrigens, bin ab jetzt in Pension
Gratuliere Dir.

-
Cool, App läuft einwandfrei!

-
Hallo,
habe bei JHU die Tabelle verbreitert, damit auch die Tageswerte da sind und habe Süd-Korea repariert (da hier ein Fehler in den Daten ist)
-
RKI hat neue Werte hinzugefügt. RefDatum (da wo die Erkrankung aufgetreten ist) und Zahlen für genesen:
NOW=`date +"%d.%m.%g %H:%M.%S"` NOWDAT=`date +"%d_%m_%g"` USER=DBUSER PASS=DBPASSWORD rm /var/skripte/data/cor*.csv #wget -O /var/skripte/data/cor_rki.csv https://opendata.arcgis.com/datasets/dd4580c810204019a7b8eb3e0b329dd6_0.csv python3 -u /var/skripte/rkijson.py wget -O /var/skripte/data/cor_landkreise.csv https://opendata.arcgis.com/datasets/917fc37a709542548cc3be077a786c17_0.csv wget -O /var/skripte/data/cor_bundesland.csv https://opendata.arcgis.com/datasets/ef4b445a53c1406892257fe63129a8ea_0.csv cp /var/skripte/data/cor_rki.csv /var/skripte/data/rki_$NOWDAT.csv.backup cp /var/skripte/data/cor_landkreise.csv /var/skripte/data/landkreise_$NOWDAT.csv.backup cp /var/skripte/data/cor_bundesland.csv /var/skripte/data/bundesland_$NOWDAT.csv.backup #mysql -u $USER -p$PASS iobroker < /var/skripte/data/createTable.txt mysqlimport --fields-terminated-by=, --ignore-lines=1 --verbose --delete --local -u $USER -p$PASS iobroker /var/skripte/data/cor_rki.csv mysqlimport --fields-terminated-by=, --ignore-lines=1 --verbose --delete --local -u $USER -p$PASS iobroker /var/skripte/data/cor_landkreise.csv mysqlimport --fields-terminated-by=, --ignore-lines=1 --verbose --delete --local -u $USER -p$PASS iobroker /var/skripte/data/cor_bundesland.csv mysql -u $USER -p$PASS iobroker < /var/skripte/data/createZiel.txtimport csv, json import datetime as dt import requests outfile = r'/var/skripte/data/cor_rki.csv' count = "https://services7.arcgis.com/mOBPykOjAyBO2ZKk/arcgis/rest/services/RKI_COVID19/FeatureServer/0/query?where=1%3D1&outFields=*&returnCountOnly=true&f=pjson" anz = requests.get(count) anz_json = json.loads(anz.text) anzahl = anz_json['count'] print("Zeilen:" + str(anzahl)) ofile = open(outfile, 'w+') output = csv.writer(ofile) search1 = "https://services7.arcgis.com/mOBPykOjAyBO2ZKk/arcgis/rest/services/RKI_COVID19/FeatureServer/0/query?where=1%3D1&outFields=*&resultOffset=" search2 = "&resultRecordCount=1000&f=pjson" i = 0 while i < anzahl: print("Readfile:" + str((int)(i / 1000)) + " von " + str((int)(int(anzahl) / 1000))) s_str = search1 + str(i) + search2 x = requests.get(s_str) data = json.loads(x.text) daten = data['features'] for row in daten: zeit = row['attributes']['Meldedatum'] / 1000 row['attributes']['Meldedatum'] = dt.datetime.utcfromtimestamp(zeit).strftime( "%Y-%m-%dT%H:%M:%S.000Z") meld = row['attributes']['Refdatum'] / 1000 row['attributes']['Refdatum'] = dt.datetime.utcfromtimestamp(meld).strftime( "%Y-%m-%dT%H:%M:%S.000Z") dat = row['attributes']['Datenstand'] row['attributes']['Datenstand'] = dat.replace(",",":") if i == 0: output.writerow(daten[0]['attributes'].keys()) for row in data['features']: output.writerow(row['attributes'].values()) i = i + 1000createTable.txt:
CREATE TABLE IF NOT EXISTS cor_rki( IdBundesland INTEGER NOT NULL ,Bundesland VARCHAR(44) NOT NULL ,Landkreis VARCHAR(44) NOT NULL ,Altersgruppe VARCHAR(9) NOT NULL ,Geschlecht VARCHAR(9) NOT NULL ,AnzahlFall INTEGER NOT NULL ,AnzahlTodesfall INTEGER NOT NULL ,ObjectId INTEGER NOT NULL PRIMARY KEY ,Meldedatum VARCHAR(24) NOT NULL ,IdLandkreis VARCHAR(5) NOT NULL ,Datenstand VARCHAR(22) NOT NULL ,NeuerFall INTEGER NOT NULL ,NeuerTodesfall INTEGER NOT NULL ,Refdatum VARCHAR(24) NOT NULL ,NeuGenesen INTEGER NOT NULL ,AnzahlGenesen INTEGER NOT NULL ) DEFAULT CHARACTER SET = UTF8; ALTER TABLE `cor_rki` ADD KEY `Meldedatum` (`Meldedatum`), ADD KEY `IdLandkreis` (`IdLandkreis`), ADD KEY `Datenstand` (`Datenstand`), ADD KEY `Refdatum` (`Refdatum`); COMMIT; CREATE TABLE IF NOT EXISTS cor_landkreise( OBJECTID INTEGER NOT NULL PRIMARY KEY ,ADE INTEGER ,GF INTEGER ,BSG BIT ,RS VARCHAR(5) NOT NULL ,AGS VARCHAR(5) ,SDV_RS VARCHAR(11) ,GEN VARCHAR(44) NOT NULL ,BEZ VARCHAR(44) NOT NULL ,IBZ INTEGER ,BEM VARCHAR(13) ,NBD VARCHAR(4) ,SN_L INTEGER ,SN_R INTEGER ,SN_K INTEGER ,SN_V1 INTEGER ,SN_V2 INTEGER ,SN_G INTEGER ,FK_S3 VARCHAR(1) ,NUTS VARCHAR(5) ,RS_0 INTEGER ,AGS_0 INTEGER ,WSK VARCHAR(23) ,EWZ INTEGER NOT NULL ,KFL NUMERIC(7,2) ,DEBKG_ID VARCHAR(16) ,Shape_Area NUMERIC(17,7) NOT NULL ,Shape_Length NUMERIC(17,10) NOT NULL ,death_rate NUMERIC(17,15) NOT NULL ,cases INTEGER NOT NULL ,deaths INTEGER NOT NULL ,cases_per_100k NUMERIC(17,14) NOT NULL ,cases_per_population NUMERIC(19,17) NOT NULL ,BL VARCHAR(22) NOT NULL ,BL_ID INTEGER NOT NULL ,county VARCHAR(36) NOT NULL ,last_update VARCHAR(16) NOT NULL ) DEFAULT CHARACTER SET = UTF8; ALTER TABLE `cor_landkreise` ADD KEY `RS` (`RS`); COMMIT; CREATE TABLE IF NOT EXISTS cor_bundesland( ID INTEGER NOT NULL PRIMARY KEY ,LAN_ew_AGS INTEGER NOT NULL ,LAN_ew_GEN VARCHAR(44) NOT NULL ,LAN_ew_BEZ VARCHAR(44) NOT NULL ,LAN_ew_EWZ INTEGER NOT NULL ,OBJECTID INTEGER NOT NULL ,Fallzahl INTEGER NOT NULL ,Aktualisierung VARCHAR(24) NOT NULL ,AGS_TXT INTEGER NOT NULL ,GlobalID VARCHAR(36) NOT NULL ,faelle_100000_EW NUMERIC(16,13) NOT NULL ,Shape_Area NUMERIC(17,5) NOT NULL ,Shape_Length NUMERIC(16,9) NOT NULL ,Death INTEGER NOT NULL ) DEFAULT CHARACTER SET = UTF8; truncate cor_rki; truncate cor_bundesland; truncate cor_landkreise;createZiel.txt:
Achtung falls ihr die Tabelle cor_datum nicht verwendet, dann die Bezüge hier löschen (Tabelle siehe nächste Post)DROP TABLE IF EXISTS cor_view; CREATE TABLE cor_view AS SELECT r.IDBundesLand as ID_B, r.IDLandkreis as ID_L,SUBSTRING(r.MeldeDatum,1,10) as R_MeldeDatum, r.ObjectID as ID_R, r.Bundesland as R_Bundesland, r.Landkreis as R_Landkreis, r.Altersgruppe as R_Alter, r.Geschlecht as R_Geschl, r.AnzahlFall as R_Fall, r.AnzahlTodesfall as R_Tote, r.Datenstand as R_Datenstand, r.NeuerFall as R_Neuerfall, r.NeuerTodesFall as R_NeuerTodesFall, SUBSTRING(r.Refdatum,1,10) as R_Refdatum, r.NeuGenesen as R_NeuGenesen, r.AnzahlGenesen as R_AnzahlGenesen, b.LAN_ew_EWZ as B_Einwohner,b.FallZahl as B_Fallzahl,b.Death as B_Tote, l.EWZ as L_Einwohner,l.KFL as L_Flaeche, l.death_rate as L_TodesRate, l.cases as L_Faelle, l.deaths as L_Tote, l.cases_per_100k as L_Faelle_pro_100000,l.cases_per_population as L_ Faelle_pro_Bevoelkerung, k.skreis as K_SKreis, k.bevoelkerung as K_Bevoelkerung, k.maenner as K_Maenner,k.frauen as K_Frauen, k.dichte as K_Dichte FROM cor_rki r,cor_bundesland b,cor_landkreise l, kreise k where r.IdLandkreis=l.RS and r.IdBundesland=b.id and r.IdLandkreis=k.id and r.AnzahlFall>0 order by ID_B,ID_L,R_MeldeDatum; update cor_view set R_Tote=0 where R_Tote<0; update cor_view set R_AnzahlGenesen=0 where R_AnzahlGenesen<0; update cor_datum set rki=false; update cor_datum set rki=true where d_datum<=(select max(R_meldedatum) from cor_view) and d_datum>=(select min(R_meldedatum) from cor_view); ALTER TABLE `cor_view` ADD UNIQUE KEY `PRIME` (`ID_B`,`ID_L`,`R_MeldeDatum`,`ID_R`) USING BTREE, ADD KEY `I1` (`R_MeldeDatum`,`R_Bundesland`,`R_Fall`,`R_Tote`), ADD KEY `I2` (`R_MeldeDatum`,`R_Landkreis`,`R_Fall`,`R_Tote`), ADD KEY `I3` (`R_MeldeDatum`,`K_SKreis`,`R_Fall`,`R_Tote`); COMMIT; -
Um einfacher Abfragen zu können habe ich eine Tabelle:
cor_datum angelegt, hier sind die vorhandenen Datumswerte markiert:
grafana:
ioBroker Corona-1586463967124.json
Wenn ihr im json R_meldedatum durch R_refdatum ersetz, dann bekommt ihr die Kurven nach Erkrankungsdatum und nicht nach Meldedatum.